xhca
Version 1.0
Fonctions :
xhca est un programme de tracé de diagrammes H.C.A. (Hydrophobic
Cluster Analysis) de séquences de protéines.
|
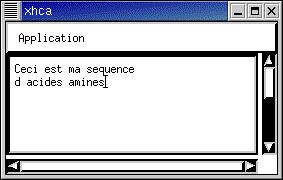
L'utilisateur entre sa séquence
dans une fenêtre en la tapant au
clavier ou par copier/coller
|
Puis il choisit un des types de tracés possibles :
-
tracé standard : (recommandé)
Dans ce cas seuls les options noir/blanc ou couleur et le titre sont
disponibles.
il est très similaire au tracé standard; seul son "look"
est un peu différent.
|
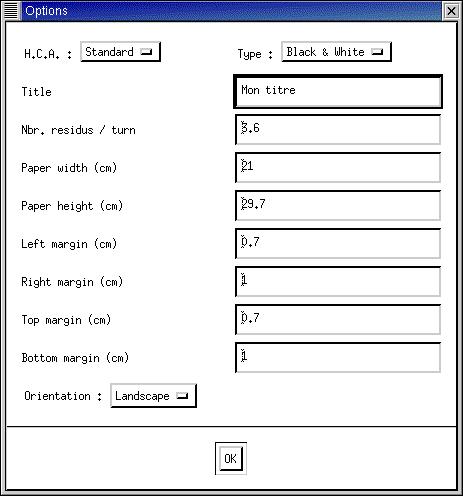
Dans ce cas des options pour
l'orientation, la taille de la feuille
de papier et les marges sont
disponibles
|
il permet en plus de choisir le nombre de résidus par tour,
pour des hélices qui ne sont pas alpha.
Dans ce cas les mosaiques ne sont pas dessinés et les Prolines
ne cassent pas les amas hydrophobes.
Enfin l'utilisateur choisit le nom du fichier pour sauvegarder
sa séquence:
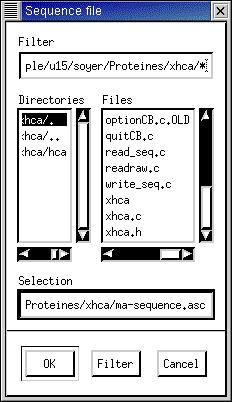
|
par exemple
ma-sequence.asc (ou .seq)
et le tracé se fera dans un
fichier en format PostScript
ma-sequence.ps
|
Ressources nécessaires :
Station de travail Unix (ou PC sous Linux, ou Mac sous MacOS 10) disposant
d'un compilateur C, et des bibliothèques X-Window et Motif.
Écran supportant X-Window ou Terminal X.
Distribution :
Les sources du programme sont disponibles librement (sans garantie) par
ftp, sous forme de fichier tar :
xhca1.0.tar.Z
depuis le réseau externe, ou
xhca1.0.tar.Z
depuis le réseau local.
Installation :
Décompresser le fichier tar : "uncompress xhca1.0.tar.Z"
en extraire les fichiers : "tar -xvf xhca1.0.tar"
dans le répertoire xhca/hca modifier le makefile pour l'adapter à votre
machine, puis l'exécuter :
"cd xhca/hca"
"vi makefile" ...
"make"
dans le répertoire xhca modifier le makefile pour l'adapter à votre
machine, puis l'exécuter :
"cd .."
"vi makefile" ...
"make"



